Diversidad genética de Capsicum pubescens mediante marcadores genómicos funcionales del CYP450
DOI:
https://doi.org/10.29312/remexca.v15i5.3733Palabras clave:
Capsicum pubescens, variabilidad genéticaResumen
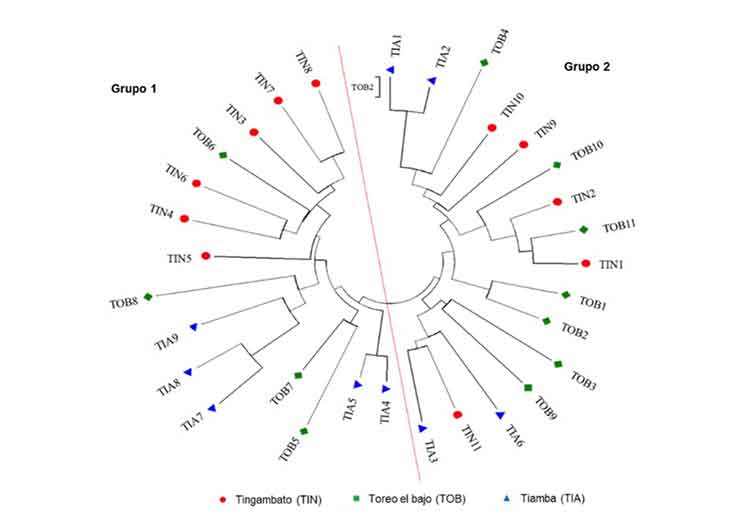
El trabajo de investigación se desarrolló en el Laboratorio de Recursos Fitogenéticos de la Facultad de Agrobiología ‘Presidente Juárez’ de la Universidad Michoacana de San Nicolás de Hidalgo, en 2019 y 2022. La investigación se planteó con el objetivo de evaluar la utilidad de los marcadores funcionales derivados del CYP450 para estudios de diversidad genética en Capsicum pubescens. El material genético consistió en 31 variedades cultivadas de C. pubescens procedentes de tres diferentes localidades del estado de Michoacán, México. El ADN genómico fue obtenido con base en el protocolo de Huang et al. (2013) y en el análisis se incluyeron dos combinaciones de cebadores del CYP450. Los productos de amplificación fueron separados en geles de acrilamida al 8% y teñidos con nitrato de plata. Un total de 85 loci fueron detectados: la combinación CYP2B6F/CYP2C19R, detectó 34 loci polimórficos; mientras que con la combinación CYP2C19F/CYP1A1R, solo 27. El análisis de diversidad de C. pubescens permitió identificar 1.54 alelos por locus, 1.33 número efectivo de alelos por locus, índice de Shannon de 0.3, un índice de heterocigosidad de 0.2 y un 60.39% de loci polimórficos. Los resultados obtenidos muestran que los marcadores derivados del CYP450, son una alternativa eficiente y de bajo costo para estudios de diversidad genética en especies vegetales.
Descargas
Citas
Aguirre, H. E. y Muñoz, O. V. 2015. El chile como alimento. Revista Ciencia. 66(3):16-23. https://www.revistaciencia.amc.edu.mx/images/revista/66-3/PDF/Chile.pdf.
Bak, S.; Beisson, F.; Bishop, G.; Hamberger, B.; Höfer, R.; Paquette, S. and Werck-Reichhart, R. D. 2011. Cytochromes P450. The Arabidopsis Book. 9:2-56. https://doi.org/10.1199/tab.0028.
Bobadilla-Larios, V.; Esparza-Ibarra, E.; Delgadillo-Ruiz, L.; Gallegos-Flores, P. y Ayala-Lujan, J. L. 2017. Variedades de chile (Capsicum annuum L.) identificadas mediante marcadores RAPD. Tropical and Subtropical Agroecosystems. 20(3):465-473. https://www.redalyc.org/pdf/939/93953814014.pdf.
Carvalho, S. I. C.; Ragassi, C. F.; Oliveira, I. B.; Amaral, Z. P. S.; Reifschneider, F. J. B.; Faleiro, F. G. and Buso, G. S. C. 2015. Transferability of microsatellite markers of Capsicum annuum L. to C. frutescens L. and C. chinense Jacq. Genetics and Molecular Research. 14(3):7937-7946. https://doi.org/10.4238/2015.July.17.1.
Castañón-Nájera, G.; Ramírez-Meraz, M.; Ruiz-Salazar, R. y Mayek-Pérez, N. 2011. Aplicación de marcadores AFLP para explorar heterosis en Capsicum spp. Revista Internacional de Botánica Experimental. 80(1):53-58. http://www.revistaphyton.fundromuloraggio.org.ar/vol80/Castanon-Najera-2011.pdf.
Contreras-Toledo, A. R.; López-Sánchez, H.; Santacruz-Varela, A.; Valadez-Moctezuma, E.; Aguilar-Rincón, V. H.; Corona-Torres, T. y López, P. A. 2011. Diversidad genética en México de variedades nativas de chile “poblano” mediante microsatélites. Revista Fitotecnia Mexicana. 34(4):225-232. http://www.scielo.org.mx/scielo.php?pid=S018773802011000400003yscript=sci-arttext.
Dorcas, J. O. F. 2011. Fundamentos de Biología Molecular. Ed. UOC. 1ra. Ed. Barcelona. 244-245 pp.
Escalera-Ordaz, A. K. y Guillén-Andrade, H. 2019. Formas y colores del chile perón. Revista Saber Más. 48(8):42-44. https://www.sabermas.umich.mx/archivo/ articulos/416-numero-48/786-chile-peronvariabilidad-de-formas-y-colores.html.
Gilani, A. S.; Kikuchi, A. and Watanabe, K. N. 2009. Genetic variation within and among fragmented populations of endangered medicinal plant, Withania coagulans (Solanaceae) from Pakistan and its implications for conservation. African Journal of Biotechnology. 8(13):2948-2958. http://www.academicjournals.org/AJB/PDF/ pdf2009/6%20Jul/Gilani%20et%20al.pdf.
Gil-Langarica, H. R. y Mayek-Pérez, N. 2008. Los marcadores moleculares en el mejoramiento genético de la resistencia a enfermedades del frijol (Phaseolus vulgaris L.): aplicaciones y perspectivas. Revista Mexicana de Fitopatología. 26(2):164-176.
Giraldo, H. P. A.; Uribe, S. S. I. y López, R. A. 2011. Análisis de secuencias de ADN mitocondrial (Cytb y ND1) en Lucilia eximia (Diptera: Calliphoridae). Revista Colombiana de Entomología. 37(2):273-278. http://www.scielo.org.co/ pdf/rcen/v37n2/v37n2a20.pdf.
Glasenapp, J. S.; Frieden, B. R. and Cruz, C. D. 2015. Shannon mutual information applied to genetic systems. Quantitative Biology. 1(1):1-20. https://arxiv.org/ftp/arxiv/papers/1512/1512.02324.pdf.
Gonzáles-Mendoza, D. 2009. El complejo enzimático citocromo p450 en las plantas. Revista internacional de contaminación ambiental. 23(4):177-183. http://www.scielo.org.mx/pdf/rica/v23n4/v23n4a3.pdf.
Guzmán, F. A.; Ayala, H. D.; Azurdia, C. A.; Duque, M. C. and De Vicente, M. C. 2005. AFLP assessment of genetic diversity of Capsicum genetic resources in Guatemala: Home gardens as an option for conservation. Crop Science. 45(1):363-370. http://qualquant.org/wp-content/uploads/ethnoecology/2005%20 Guzman363.pdf.
Helliwell, C. A.; Chandler, P. M.; Poole, A.; Dennis, E. S. and Peacock, W. J. 2000. The CYP88A cytochrome P450, ent-kaurenoic acid oxidase, catalyzes three steps of the gibberellin biosynthesis pathway. Proceedings of the National Academy of Sciences. USA. 98(4):2065- 2070.
Huang, Q. X.; Wang, X. C.; Kong, H.; Guo, Y. L. and Guo, A. P. 2013. An efficient DNA isolation method for tropical plants. African Journal of Biotechnology. 12(19):2727-2732. https://doi.org/10.5897/AJB12.524.
Inui, H.; Kodama, T.; Ohkawa, Y. and Ohkawa, H. 2000. Herbicide metabolism and cross tolerance in transgenic potato plants co-expressing human CYP1A1, CYP2B6, and CYP2C19. Pesticide Biochemistry and Physiology. 66(2):116-129.
Irwin, D. M.; Kocher, T. D. and Wilson, A. C. 1991. Evolution of the cytochrome b gene of mammals. Journal of Molecular Evolution. 32(2):128-144.
Jaimes-Santoyo, J.; Montesinos-Sampedro, A.; Barbosa-Cobos, R. E.; Moreno-Mutio, S. G.; Rodríguez-Ballesteros, D.; Ramos-Cervantes, T.; Ocharán-Hernández, M. E.; Toscano-Garibay, J. y Beltrán-Ramírez, O. 2014. El citocromo P-450. Revista del Hospital Juárez de México. 81(4):250-256. https://www.medigraphic.com/pdfs/juarez/ju-2014/ju144j.pdf.
Li, G. and Quiros, C. F. 2001. Sequence-related amplified polymorphism (SRAP) a new marker system based on a simple PCR reaction: its application to mapping and gene tagging in brassica. Genética Teórica y Aplicada. 103(2-3):455-461.
Lijun, O. and Xuexiao, Z. 2012. Inter simple sequence repeat analysis of genetic diversity of five cultivated pepper species. African Journal of Biotechnology. 11(4):752-757. https://doi.org/10.5897/AJB10.2551.
López-Espinosa, S. T.; Latournerie-Moreno, L.; Castañón-Nájera, G.; Ruiz-Sánchez, E.; Gómez-Leyva, J. F.; Andueza-Noh, R. H. y Mijangos-Cortés, J. O. 2018. Diversidad genética de chile habanero (Capsicum chinense jacq.) mediante ISSR. Revista Fitotecnia Mexicana. 41(3):227-236. https://doi.org/10.35196/rfm.2018.3.227-236.
Machida-Hirano, R.; Cortés-Cruz, M.; González, B. A. A.; Íñiguez, J. C.; Shirata, K. and Watanabe, K. N. 2015. Isolation and characterization of novel microsatellite markers in chayote [Sechium edule (Jacq.) Sw.]. American Journal of Plant Sciences. 6(13):2033-2041. https://doi.org/10.4236/ajps.2015.613203.
Mahmoud, A. S. 2013. Inter-simple sequence repeat (ISSR) markers in the evaluation of genetic polymorphism of Egyptian Capsicum L. hybrids. African Journal of Biotechnology. 12(7):665-669.
Martínez, C. L. 2007. Reconstrucción de la historia de cambio de los caracteres. Ed. Ecología Molecular. 87-152 pp. https://www.researchgate.net/publication/ 258129643-Ecologia-Molecular.
Panwar, B.; Saini, R. K.; Sharma, N.; Yadav, D. and Kumar, A. 2010. Efficiency of RAPD, SSR and Cytochrome P450 gene-based markers in accessing genetic variability amongst finger millet (Eleusine coracana) accessions. Molecular Biology Reports. https://doi.org/10.1007/s11033-010-0067-5.
Pardey, R. C. y García, D. M. A. 2011. Caracterización molecular de 135 introducciones de Capsicum procedentes del banco de germoplasma de la Universidad Nacional de Colombia sede Palmira. Revista Intropica. 6(1):21-32. https://agris.fao.org/agris-search/search.do?recordID=DJ20220180543.
Peakall, R. and Smouse, P. E. 2012. GenAlex 6.5: Genetic analysis in Excel. Population genetic software for teaching and research-an update. Bioinformatics. 28(19):2537-2539. https://doi.org/10.1093/bioinformatics/bts460.
Rentería, A. M. 2007. Breve revisión de los marcadores moleculares. Ed. Ecología Molecular. 541-571. https://hopelchen.tecnm.mx/principal/sylabus/fpdb/ recursos/r119349.PDF.
Ríos, E.; Mejía-Ruiz, H. y Álvarez-Castañeda, S. T. 2009. Marcadores moleculares: una revolución en la Zoología. Revista Ciencia. 60(3):5-13.
Saini, R. K.; Saad, K. R.; Ravishankar, G. A.; Giridhar, P. and Shetty, N. P. 2013. Genetic diversity of commercially grown Moringa oleifera Lam. cultivars from India by RAPD, ISSR and cytochrome P 450-based markers. Plant Systematics and Evolution. 299(7):1205-1213. https://doi.org/10.1007/s00606-013-0789-7.
Sanguinetti, C. J. and Simpson, A. J. 1994. Rapid silver staining and recovery of PCR products separated on polyacrylamide gels. Biotechniques. 17(5):914-921.
Shakeel, A. J.; Kikuchi, A.; Ahmad, D. and Watanabe, K. N. 2019. Characterization of the genetic structure of mango ginger (Curcuma amada Roxb.) from Myanmar in farm and genebank collection by the neutral and functional genomic markers. Electronic Journal of Biotechnology. 13(6):1-11. ISSN: 0717-3458.
Tamura, K.; Peterson, D.; Peterson, N.; Stecher, G.; Nei, M. and Kumar, S. 2011. MEGA5: molecular evolutionary genetics analysis using maximum likelihood, evolutionary distance, and maximum parsimony methods. Molecular Biology and Evolution. 28(10):2731-2739. https://doi.org/10.1093/molbev/msr121.
Toledo-Aguilar, R.; López-Sánchez, H.; Santacruz-Varela, A.; Valadez-Moctezuma, E.; López, P. A.; Aguilar-Rincón, V. H.; González-Hernández, V. A. and Vaquera-Huerta, H. 2016. Characterization of genetic diversity of native ‘Ancho’ chili populations of Mexico using microsatellite markers. Chilean Journal of Agricultural Research. 76(1):18-26. https://doi.org/10.4067/S0718-58392016000100003.
Valencia-Quintana, R.; Sánchez-Alarcón, J.; Gómez-Arroyo, S.; Gómez-Olivares, J. L. y Kubiak, S. M. W. 2009. Los citocromos P450 en los 5 reinos de Margulis. Ciencia en la frontera: revista de ciencia y tecnología de la UACJ. 7(1):9-26.
Vos, P.; Hogers, R.; Bleeker, M.; Reijans, M.; Lee, T.; Van De, Hornes, M. Friters, A.; Pot, J; Paleman, J.; Kuiper, M. and Zabeau, M. 1995. AFLP: A new technique for DNA fingerprinting. Nucleic Acids Research. 23(21):4407-4414. https://doi.org/10.1093/nar/23.21.4407.
Wan, Y.; Watanabe, J. A.; San, S. Y.; Htaik, T.; Win, K.; Yamanaka, S.; Nakamura, I. and Watanabe, K. N. 2005. Assessment of genetic diversity among the major Myanmar banana landraces. Breeding Science. 55(3):365-369. https://www.jstage.jst.go.jp/article/jsbbs/55/3/55-3-365/-pdf.
Whitbred, J. M. and Schuler, M. A. 2000. Molecular characterization of CYP73A9 and CYP82A1 P450 genes involved in plant defense in pea. Plant Physiol. 124(1):47-58.
Winkler, R. G. and Helentjaris, T. 1995. The maize Dwarf3 gene encodes a cytochrome P450- mediated early step in gibberellin biosynthesis. Plant Cell. 7(1):1307-1317.
Xiao-min, Z.; Zheng-hai, Z.; Xiao-zhen, G.; Sheng-li, M.; Xi-xiang, L.; Alain, J. C. P.; Li-Hao, W. and Bao-xi, Z. 2016. Genetic diversity of pepper (Capsicum spp.) germplasm resources in China reflects selection for cultivar types and spatial distribution. Journal of Integrative Agriculture. 15(9):1991-2001.
Yamanaka, S.; Suzuki, E.; Tanaka, M.; Takeda, Y.; Watanabe, J. A. and Watanabe, K. N. 2003. Assessment of cytochrome P450 sequences offers a useful tool for determining genetic diversity in higher plant species. Theoretical and Applied Genetics. 108(1):1-9. https://doi.org/10.1007/s00122-003-1403-0.
Descargas
Publicado
Cómo citar
Número
Sección
Licencia
Derechos de autor 2024 Revista Mexicana de Ciencias Agrícolas

Esta obra está bajo una licencia internacional Creative Commons Atribución-NoComercial 4.0.
Los autores(as) que publiquen en Revista Mexicana de Ciencias Agrícolas aceptan las siguientes condiciones:
De acuerdo con la legislación de derechos de autor, Revista Mexicana de Ciencias Agrícolas reconoce y respeta el derecho moral de los autores(as), así como la titularidad del derecho patrimonial, el cual será cedido a la revista para su difusión en acceso abierto.
Los autores(as) deben de pagar una cuota por recepción de artículos antes de pasar por dictamen editorial. En caso de que la colaboración sea aceptada, el autor debe de parar la traducción de su texto al inglés.
Todos los textos publicados por Revista Mexicana de Ciencias Agrícolas -sin excepción- se distribuyen amparados bajo la licencia Creative Commons 4.0 atribución-no comercial (CC BY-NC 4.0 internacional), que permite a terceros utilizar lo publicado siempre que mencionen la autoría del trabajo y a la primera publicación en esta revista.
Los autores/as pueden realizar otros acuerdos contractuales independientes y adicionales para la distribución no exclusiva de la versión del artículo publicado en Revista Mexicana de Ciencias Agrícolas (por ejemplo incluirlo en un repositorio institucional o darlo a conocer en otros medios en papel o electrónicos) siempre que indique clara y explícitamente que el trabajo se publicó por primera vez en Revista Mexicana de Ciencias Agrícolas.
Para todo lo anterior, los autores(as) deben remitir el formato de carta-cesión de la propiedad de los derechos de la primera publicación debidamente requisitado y firmado por los autores(as). Este formato debe ser remitido en archivo PDF al correo: revista_atm@yahoo.com.mx; revistaagricola@inifap.gob.mx.
Esta obra está bajo una licencia de Creative Commons Reconocimiento-No Comercial 4.0 Internacional.


