Detección del gen xyl3 en cepas de Fusarium oxysporum f. sp. vanillae
DOI:
https://doi.org/10.29312/remexca.v14i6.2711Palabras clave:
gen xilanasa, mutaciones, selección positivaResumen
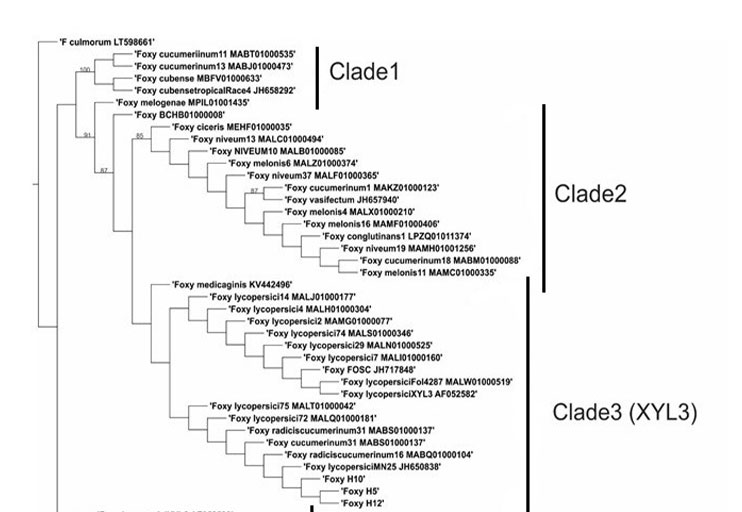
Los mecanismos de Fusarium oxysporum relacionados con la degradación de componentes estructurales de la raíz, como el xilano, son muy importantes, dado que la colonización de este órgano es pieza clave en el establecimiento de la enfermedad. El presente estudio se enfocó en detectar el gen codificante para la enzima xilanasa xyl3 en cepas de F. oxysporum f. sp. vanillae y buscar homólogos a este gen en secuencias de otras formae speciales y especies del género Fusarium, con el fin de determinar las relaciones filogenéticas entre las xilanasas dentro del complejo de especies F. oxysporum, así como, buscar evidencia de selección natural en el año 2020. Los resultados indicaron que, de las nueve cepas evaluadas, solo tres tuvieron una copia del gen xyl3. La filogenia mostró ocho clados, donde el clado 3 fue consistente con la clasificación de xyl3, mientras que los otros tipos de xilanasas quedaron agrupados en el clado 2. La prueba de selección natural no mostró evidencia de selección positiva dentro de la filogenia, lo que sugiere que la mutación neutral es la responsable de la diversidad en el gen xilanasa entre el complejo de especies F. oxysporum, lo que lleva a proponer que el gen no parece haber cambiado con la colonización de nuevos hospedantes.
Descargas
Citas
Abdul-Karim, N. F.; Mohd, M.; Izham-Mohd, N. M and Zakaria, L. 2016. Saprophytic and potentially pathogenic Fusarium species from peat soil in Perak and Pahang. Trop Life Sci. Res. 27(1):1-20. https://www.ncbi.nlm.nih.gov/pmc/articles/PMC4807956/.
Adame-García, J.; Trigos, Á.; Iglesias-Andreu, L. G.; Flores-Estévez, N. and Luna-Rodríguez, M. 2011. Isozymic and pathogenic variations of Fusarium spp. associated with vanilla stem and root rotting. Trop subtrop agroecosystems. 13(3):299-306. https://www.revista.ccba.uady.mx/ojs/index.php/TSA/article/view/1330/663.
Adame-García, J.; Rodriguez-Guerra, R.; Iglesias-Andreu, L. G.; Ramos-Prado, J. M. and Luna-Rodríguez, M. 2015. Molecular identification and pathogenic variation of Fusarium species isolated from Vanilla planifolia in Papantla Mexico. Bot. Sci. 93(3):669-678. https://doi.org/10.17129/botsci.142. DOI: https://doi.org/10.17129/botsci.142
Adame-García, J.; Flores-Rosa, F. R.; Ricaño-Rodríguez, J. and Luna-Rodríguez, M. 2016. Adequacy of a protocol for amplification of EF-1α gene of Fusarium oxysporum f. sp. vanillae. ARPN J. Agric. Biol. Sci. 11(6):236-241. http://www.arpnjournals.org/jabs/ research-papers/rp-2016/jabs-0616-804.pdf.
Demers, J. E.; Gugino, B. K. and Jiménez-Gasco, M. M. 2015. Highly diverse endophytic and soil Fusarium oxysporum populations associated with field-grown tomato plants. Appl. Environ. Microbiol. 81(1):81-90. https://doi.org/10.1128/AEM.02590-14. DOI: https://doi.org/10.1128/AEM.02590-14
De-Vries, R. P. and Visser J. 2001. Aspergillus enzymes involved in degradation of plant cell wall polysaccharides. Microbiol Mol. Biol Rev. 65(4):497-522. https://doi.org/10.1128/MMBR.65.4.497-522.2001. DOI: https://doi.org/10.1128/MMBR.65.4.497-522.2001
Edel-Hermann, V. and Lecomte, C. 2019. Current status of Fusarium oxysporum formae speciales and races. Phytopathology. 109(4):512-530. https://doi.org/10.1094/PHYTO-08-18-0320-RVW.
Flores-Rosa, F. R.; Luna, E.; Adame-García, J.; Iglesias-Andreu, L. G. and Luna-Rodríguez, M. 2018. Phylogenetic position and nucleotide diversity of Fusarium oxysporum f. sp. vanillae worldwide based on translation elongation factor 1α sequences. Plant Pathol. 67(6):1278-1285. https://doi.org/10.1111/ppa.12847. DOI: https://doi.org/10.1111/ppa.12847
Goloboff, P. A.; Farris, J. S. and Nixon, K. C. 2008. TNT, a free program for phylogenetic analysis. Cladistics. 24(5):774-786. https://doi.org/10.1111/j.1096-0031.2008.00217.x. DOI: https://doi.org/10.1111/j.1096-0031.2008.00217.x
González-Oviedo, N.; Iglesias-Andreu, L. G.; Flores-Rosa, F. R.; Rivera-Fernández, A. and Luna-Rodríguez M. 2022. Genetic analysis of the fungicide resistance in Fusarium oxysporum associated to Vanilla planifolia. Mex. J. Phytopathol. 40(3):1-19. https://doi.org/10.18781/R.MEX.FIT.2203-3.
Gómez-Gómez, E.; Ruíz-Roldán, M. C.; Pietro, A.; Roncero, M. I. G. and Hera, C. 2002. Role in pathogenesis of two endo-β-1,4-xylanase genes from the vascular wilt fungus Fusarium oxysporum. Fungal Genet Biol. 35(3):213-222. https://doi.org/10.1006/ fgbi.2001.1318. DOI: https://doi.org/10.1006/fgbi.2001.1318
Gurjar, G.; Barve, M.; Giri, A. and Gupta, V. 2009. Identification of Indian pathogenic races of Fusarium oxysporum f. sp. ciceris with gene specific, ITS and random markers. Mycologia. 101(4):484-495. https://doi.org/10.3852/08-085. DOI: https://doi.org/10.3852/08-085
Hall, T A. 1999. BioEdit: a user-friendly biological sequence alignment editor and analysis program for Windows 95/98/NT. Nucleic Acids Symp Ser. 41:95-98.
Hughes, A. L. and Friedman, R. 2008. Codon-based tests of positive selection, branch lengths, and the evolution of mammalian immune system genes. Immunogenetics. 60:495-506. https://doi.org/10.1007/s00251-008-0304-4. DOI: https://doi.org/10.1007/s00251-008-0304-4
Jorge, I.; Rosa, O.; Navas-Cortés, J. A.; Jiménez-Díaz, R. M and Tena, M. 2005. Extracellular xylanases from two pathogenic races of Fusarium oxysporum f. sp. ciceris: enzyme production in culture and purification and characterization of a major isoform as an alkaline endo beta xylanase of low molecular weight. Antonie van Leeuwenhoek. 88:48-59. https://doi.org/10.1007/s10482-004-7584-y. DOI: https://doi.org/10.1007/s10482-004-7584-y
Kalluri, U. C.; Yin, H.; Yang, X. and Davison, B. H. 2014. Systems and synthetic biology approaches to alter plant cell walls and reduce biomass recalcitrance. Plant Biotechnol J. 12(9):1207-1216. https://doi.org/10.1111/pbi.12283. DOI: https://doi.org/10.1111/pbi.12283
Koyyappurath, S.; Conéjéro, G.; Dijoux, J. B.; Lapeyre-Montès, F.; Jade, K.; Chiroleu, F.; Gatineau, F.; Verdeil, J. L.; Besse, P. and Grisoni, M. 2015. Differential responses of vanilla accessions to root rot and colonization by Fusarium oxysporum f. sp. radicis-vanillae. Front Plant Sci. 6:1-16. https://doi.org/10.3389/fpls.2015.01125. DOI: https://doi.org/10.3389/fpls.2015.01125
Koyyappurath, S.; Atuahiva, T.; Le Guen, R.; Batina, H.; Le Squin, S.; Gautheron, N.; Edel-Hermann, V.; Peribe, J.; Jahiel, M.; Steinberg, C.; Liew, E. C. Y.; Alabouvette, C.; Besse, P.; Dron, M.; Sache, I.; Laval, V. and Grisoni, M. 2016. Fusarium oxysporum f. sp. radicis-vanillae is the causal agent of root and stem rot of vanilla. Plant Pathol. 65(4):612-625. https://doi.org/10.1111/ppa.12445. DOI: https://doi.org/10.1111/ppa.12445
Kumar, S.; Stecher, G. and Tamura, K. 2016. MEGA7: molecular evolutionary genetics analysis version 7.0 for bigger datasets. Mol. Biol. Evol. 33(7):1870-1874. https://doi.org/10.1093/molbev/msw054. DOI: https://doi.org/10.1093/molbev/msw054
Laurence, M. H.; Summerell, B. A. and Liew, E. C. Y. 2015. Fusarium oxysporum f. sp. canariensis: evidence for horizontal gene transfer of putative pathogenicity genes. Plant Pathol. 64(5):1068-75. https://doi.org/10.1111/ppa.12350. DOI: https://doi.org/10.1111/ppa.12350
Metzger, K. J. and Thomas, M. A. 2010. Evidence of positive selection at codon sites localized in extracellular domains of mammalian CC motif chemokine receptor proteins. BMC Evol. Biol. 10(139):1-9. https://doi.org/10.1186/1471-2148-10-139. DOI: https://doi.org/10.1186/1471-2148-10-139
Nei, M. and Gojobori, T. 1986. Simple methods for estimating the numbers of synonymous and nonsynonymous nucleotide substitutions. Mol. Biol. Evol. 3(5):418-426. https://doi.org/10.1093/oxfordjournals.molbev.a040410. DOI: https://doi.org/10.1093/oxfordjournals.molbev.a040410
O’Donnell, K.; Gueidan, C.; Sink, S.; Johnston, P. R.; Crous, P. W.; Glenn, A.; Riley, R.; Zitomer, N. C.; Colyer, P. and Waalwijk, C. 2009. A two-locus DNA sequence database for typing plant and human pathogens within the Fusarium oxysporum species complex. Fungal Gen. Biol. 46(12):936-948. https://doi.org/10.1016/j.fgb.2009.08.006. DOI: https://doi.org/10.1016/j.fgb.2009.08.006
Olivain, C.; Humbert, C.; Nahalkova, J.; Fatehi, J.; L’Haridon, F. and Alabouvette, C. 2006. Colonization of tomato root by pathogenic and nonpathogenic Fusarium oxysporum strains inoculated together and separately into the soil. Appl. Environ. Microbiol. 72(2):1523-1531. https://doi.org/10.1128/AEM.72.2.1523-1531.2006. DOI: https://doi.org/10.1128/AEM.72.2.1523-1531.2006
Pattathil, S.; Hahn, M. G.; Dale, B. E. and Chundawat, S. P. S. 2015. Insights into plant cell wall structure, architecture, and integrity using glycome profiling of native and AFEXTM-pretreated biomass. J. Exp. Bot. 66(14):4279-4294. https://doi.org/10.1093/jxb/ erv107. DOI: https://doi.org/10.1093/jxb/erv107
Peña, M. J.; Kulkarni, A. R.; Backe, J. Boyd, M. O.; Neill, M. A. and York, W. S. 2016. Structural diversity of xylans in the cell walls of monocots. Planta. 244:589-606. https://doi.org/10.1007/s00425-016-2527-1. DOI: https://doi.org/10.1007/s00425-016-2527-1
Pinaria, A. G.; Liew, E. C. Y. and Burgess, L. W. 2010. Fusarium species associated with vanilla stem rot in Indonesia. Australasian Plant. Pathol. 39:176-183. https://doi.org/10.1071/AP09079. DOI: https://doi.org/10.1071/AP09079
Pinaria, A. G.; Laurence, M. H.; Burgess, L. W. and Liew, E. C. Y. 2015. Phylogeny and origin of Fusarium oxysporum f. sp. vanillae in indonesia. Plant Pathol. 64(6):1358-65. https://doi.org/10.1111/ppa.12365. DOI: https://doi.org/10.1111/ppa.12365
Ruiz-Roldan, M. C.; Di-Pietro, A.; Huertas-Gonzalez, M. D. and Roncero, M. I. 1999. Two xylanase genes of the vascular wilt pathogen Fusarium oxysporum are differentially expressed during infection of tomato plants. Mol. Gen. Genet. 261:530-536. https://doi.org/10.1007/s004380050997. DOI: https://doi.org/10.1007/s004380050997
Soto-Arenas, M. A y Solano-Gómez, A. R. 2007. Ficha técnica de Vanilla planifolia. En: información actualizada sobre las especies de orquídeas del PROY-NOM-059-ECOL-2000. Instituto Chinoin A.C. Herbario de la Asociación Mexicana de Orquideología AC. Proyecto No. W029. México. DF. 1-18 pp. http://www.conabio.gob.mx/conocimiento/ ise/fichasnom/Vanillaplanifolia00.pdf.
Turrà, D.; Ghalid, M.; Rossi, F. and Pietro, A. 2015. Fungal pathogen uses sex pheromone receptor for chemotropic sensing of host plant signals. Nature. 527:521-524. https://doi.org/10.1038/nature15516. DOI: https://doi.org/10.1038/nature15516
Waweru, B.; Turoop, L.; Kahangi, E.; Coyne, D. and Dubois, T. 2014. Non-pathogenic Fusarium oxysporum endophytes provide field control of nematodes, improving yield of banana (Musa sp.). Biological Control. 74:82-88. https://doi.org/10.1016/j.biocontrol. 2014.04.002. DOI: https://doi.org/10.1016/j.biocontrol.2014.04.002
Zhang, J.; Nielsen, R.; and Yang, Z. 2005. Evaluation of an improved branch-site likelihood method for detecting positive selection at the molecular level. Mol. Biol. Evol. 22(12):2472-2479. https://doi.org/10.1093/molbev/msi237. DOI: https://doi.org/10.1093/molbev/msi237
Descargas
Publicado
Cómo citar
Número
Sección
Licencia
Derechos de autor 2023 Revista Mexicana de Ciencias Agrícolas

Esta obra está bajo una licencia internacional Creative Commons Atribución-NoComercial 4.0.
Los autores(as) que publiquen en Revista Mexicana de Ciencias Agrícolas aceptan las siguientes condiciones:
De acuerdo con la legislación de derechos de autor, Revista Mexicana de Ciencias Agrícolas reconoce y respeta el derecho moral de los autores(as), así como la titularidad del derecho patrimonial, el cual será cedido a la revista para su difusión en acceso abierto.
Los autores(as) deben de pagar una cuota por recepción de artículos antes de pasar por dictamen editorial. En caso de que la colaboración sea aceptada, el autor debe de parar la traducción de su texto al inglés.
Todos los textos publicados por Revista Mexicana de Ciencias Agrícolas -sin excepción- se distribuyen amparados bajo la licencia Creative Commons 4.0 atribución-no comercial (CC BY-NC 4.0 internacional), que permite a terceros utilizar lo publicado siempre que mencionen la autoría del trabajo y a la primera publicación en esta revista.
Los autores/as pueden realizar otros acuerdos contractuales independientes y adicionales para la distribución no exclusiva de la versión del artículo publicado en Revista Mexicana de Ciencias Agrícolas (por ejemplo incluirlo en un repositorio institucional o darlo a conocer en otros medios en papel o electrónicos) siempre que indique clara y explícitamente que el trabajo se publicó por primera vez en Revista Mexicana de Ciencias Agrícolas.
Para todo lo anterior, los autores(as) deben remitir el formato de carta-cesión de la propiedad de los derechos de la primera publicación debidamente requisitado y firmado por los autores(as). Este formato debe ser remitido en archivo PDF al correo: revista_atm@yahoo.com.mx; revistaagricola@inifap.gob.mx.
Esta obra está bajo una licencia de Creative Commons Reconocimiento-No Comercial 4.0 Internacional.


