Estimación de Podosphaera xanthii en pepino: técnicas de aprendizaje automático con imágenes digitales
DOI:
https://doi.org/10.29312/remexca.v16i30.4039Palabras clave:
algoritmo, cenicilla, enfermedadResumen
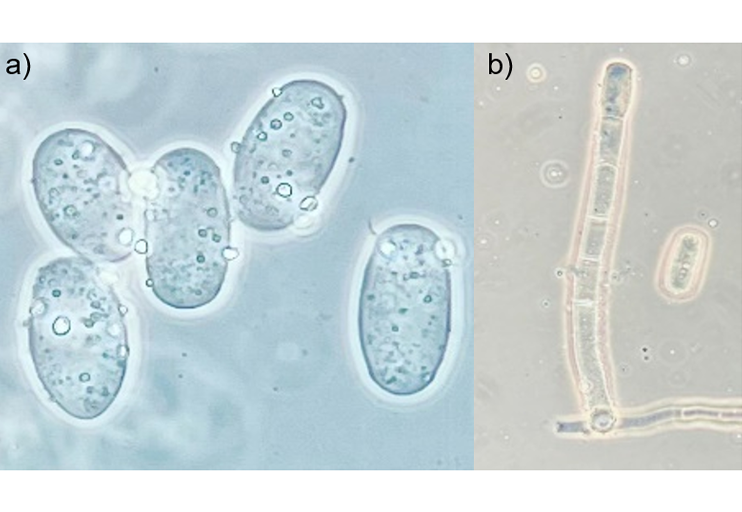
Los hongos fitopatógenos representan una amenaza considerable para el cultivo de cucurbitáceas, por lo que la detección temprana y cuantificación precisa de enfermedades resulta esencial para reducir pérdidas en la producción. En este estudio se desarrolló una metodología para estimar cuantitativamente el daño provocado por Podosphaera xanthii en hojas de pepino, mediante el uso de imágenes digitales y técnicas de aprendizaje automático. Se emplearon redes neuronales convolucionales para clasificar visualmente el grado de severidad en seis categorías predefinidas, utilizando secciones de hojas con síntomas evidentes del hongo. Adicionalmente, se entrenaron y compararon cuatro algoritmos de clasificación supervisada: K-NN, árboles de decisión, bosques aleatorios y redes neuronales. El modelo que obtuvo el mejor desempeño fue el de bosques aleatorios, con una precisión del 90%, mientras que K-NN alcanzó el valor más bajo (79%). Estos resultados posicionan al modelo como una herramienta útil para el monitoreo automatizado de enfermedades en campo, facilitando la toma de decisiones fitosanitarias. Además, la metodología ofrece una base sólida para investigadores interesados en diseñar e implementar sistemas automáticos de clasificación de enfermedades en plantas, brindando información clara sobre el rendimiento de distintas arquitecturas de clasificación. El algoritmo desarrollado en R permite adaptar y escalar esta solución a diferentes condiciones de cultivo y tipos de enfermedades foliares.
Descargas
Citas
Alaminos, F. A. F. 2023. Árboles de decisión en R con Random Forest. Obts Ciencia Abierta Alicante: limecop. 47-50 pp.
Benali, L.; Notton, G.; Fouilloy, A.; Voyant, C.; Dizene, R.; Boum, H.; Ene, E.; Alia, E.; Ezzouar, B. and Algiers, A. 2019. Solar radiation forecasting using artificial neural network and random forest methods: application to normal beam, horizontal diffuse and global components. Renewable Energy. 132:871-884. https://doi.org/10.1016/j.renene.2018.08.044.
Braun, U. and Cook, A. R. T. 2012. Taxonomic manual of the erysiphales (powdery mildews). Centraalbureau voor Schimmelcultures. 11. CBS Biodiversity: 86-644 pp.
Cipriano, G. R. y González, D. 2022. Identificación molecular de los tipos de compatibilidad en poblaciones de Podosphaera xanthii (Erysiphaceae) infectando cucurbitáceas en Veracruz, México. Acta Botánica Mexicana. 128(129):1-11. https://doi.org/10.21829/abm129.2022.2068.
Cruz, S. H.; Sanchez, M. G.; Rivera, C. J. P. and Avila, G. H. 2020. Identification of phenological stages of sugarcane cultivation using Sentinel-2 images. applications in software engineering proceedings of the 9th. International Conference on Software Process Improvement, CIMPS. 110-116 pp. https://doi.org/10.1109/CIMPS52057.2020.9390095.
Demirovi´c, D. and Stuckey, P. J. 2021. Optimal decision trees for nonlinear metrics. 3733-3744. www.aaai.org.
Fernández, A. 2023. Árboles de decisión en R con Random Forest. Obets Ciencia Abierta. Alicante: limencop. 134 p.
Figueredo, A. A. y Ballesteros, R. J. 2016. Identificación del estado de madurez de las frutas con redes neuronales artificiales, una revisión. 13(1):117-132. https://www.redalyc.org/journal/5600/560062814010/html/.
Flores, P. G.; López, I. F.; Kemp, P. D.; Dörner, J. y Zhang, B. 2016. Modelo de árbol de decisión: una herramienta para el manejo de la pradera. Agro Sur. 44(2):3-10. https://doi.org/10.4206/agrosur.2016.v44n2-02.
Ghawi, R. and Pfeffer, J. 2019. Efficient hyperparameter tuning with grid search for text categorization using knn approach with BM25 similarity. Open Computer Science. 9(1):160-180. https://doi.org/10.1515/COMP-2019-0011/machinereadablecitation/ris.
Guaillazaca, G. C. A. y Hernández, A. V. 2020. Clasificador de productos agrícolas para control de calidad basado en machine learning e industria 4.0. Revista Perspectivas. 2(2):21-28. https://doi.org/10.47187/perspectivas.vol2iss2.pp21-28.2020.
Hassoun, M. H. 1995. Fundamentals of artificial neural networks book. MIT Press. Cambridge, Massachusetts. 417-452 pp. https://kupdf.net/download/48375906-fundamentals-of-artificial-neural-networks-book-1-598b1ef3dc0d601b67300d18-pdf.
Hernández, F. Y.; González, Z. E.; Marrero, T. A. y Dueñas, G. M. J. 2007. Uso de escala para determinar severidad de enfermedades fungosas en híbridos de pepino bajo cultivo protegido. INIFAT. 11(31):49-51.
Kaushik, H.; Khanna, A.; Singh, D.; Kaur, M. and Lee, H. N. 2023. TomFusioNet: a tomato crop analysis framework for mobile applications using the multi-objective optimization based late fusion of deep models and background elimination. Applied Soft Computing. 133:1-24. https://doi.org/10.1016/j.asoc.2022.109898.
Larijani, M. R.; Asli-Ardeh, E. A.; Kozegar, E. and Loni, R. 2019. Evaluation of image processing technique in identifying rice blast disease in field conditions based on KNN algorithm improvement by K-means. Food Science and Nutrition. 7(12):3922-3930. https://doi.org/10.1002/FSN3.1251.
Li, S.; Li, K.; Qiao, Y. and Zhang, L. 2022. A multi-scale cucumber disease detection method in natural scenes based on YOLOv5. Computers and Electronics in Agricultura. 202:1-12. https://doi.org/10.1016/J.COMPAG.2022.107363.
Ma, J.; Du, K.; Zheng, F.; Zhang, L.; Gong, Z. and Sun, Z. 2018. A recognition method for cucumber diseases using leaf symptom images based on deep convolutional neural network. 154:1-7. https://doi.org/10.1016/j.compag.2018.08.048.
Mohamed, Y. F.; Bardin, M. N. P. C. and Pitrat, I. 1995. Causal agents of powdery mildew of cucubits in Sudan. Plant Disease. 79(6):635-636. https://www.apsnet.org/publications/plantdisease/backissues/Documents/1995Articles/PlantDisease79n06-634.
Morejón, G. N.; Coca, M. B. y Martínez, I. D. 2010. Mildiu polvoriento en las cucurbitáceas. Revista de Protección Vegetal. 25(1):44-50. https://revistas.censa.edu.cu/index.php/RPV/article/view/282.
Olivares, B. O.; Vega, A.; Angélica, M.; Calderón, R.; Rey, J. C. and Lobo, D. 2021. Classification of areas affected by banana wilt: an application with machine learning algorithms in Venezuela. https://revistas.up.ac.pa/index.php/REICTORCID.
OTSU, N. A. 1978. Threshold selection method from gray level histogram. IEEE. Transactions on Systems, Man and Cybernetycs. 9(1):62-66.
Pacciorett, P. A.; Kurina, F. G. y Balzarini, M. G. 2020. Muestreo de sitios a escala regional para mapeo digital basado en propiedades de suelo. Ciencia del Suelo. 38(2):310-320. https://www.scielo.org.ar/scielo.php?script=sciarttext&pid=S185020672020000200310&lng=es&tlng=es.
Paymode, A. S. and Malode, V. B. 2022. Transfer learning for multi-crop leaf disease image classification using convolutional neural Network VGG. Artificial Intelligence in Agriculture. 6:23-33. https://doi.org/10.1016/j.aiia.2021.12.002.
Ramos, R. T. V.; Castillo, A. P. J.; Ticona, J. B. y Velasco, B. J. G. 2023. Predicción del éxito del telemarketing bancario mediante el uso de árboles de decisión. Innovación y Software. 4(1):122-137. https://doi.org/10.48168/innosoft.s11.a84.
Rocha, J. F. L.; Reyes, D. Y.; Días, L. E.; Francisco, F. N. y Juárez, C. J. A. 2023. El mildiu polvoriento en calabaza: identificación y manejo bajo las condiciones de Tehuacán, México. Cultivos Tropicales. 44(2): https://cu-id.com/2050/v44n2e09. https://ediciones.inca.edu.cu/index.php/ediciones/article/view/1731.
Sarkar, C.; Gupta, D.; Gupta, U. and Hazarika, B. B. 2023. Leaf disease detection using machine learning and deep learning: review and challenges. Applied Soft Computing. 145:1-61. https://doi.org/10.1016/J.ASOC.2023.110534.
Suganya, D. K.; Srinivasan, P. and Bandhopadhyay, S. 2020. H2K-A robust and optimum approach for detection and classification of groundnut leaf diseases. Computers and Electronics in Agriculture. 178. https://doi.org/10.1016/j.compag.2020.105749.
Sujatha, R.; Chatterjee, J. M.; Jhanjhi, N. Z. and Brohi, S. N. 2021. Performance of deep learning vs machine learning in plant leaf disease detection. Microprocessors and Microsystems. 80:103615. https://doi.org/10.1016/J.MICPRO.2020.103615.
Sun, Z.; Hu, S. Y. and Wen, Y. 2022. Biological control of the cucumber downy mildew pathogen Pseudoperonospora cubensis. Horticulturae. 8:1-15. MDPI. https://doi.org/10.3390/horticulturae8050410.
Torgo L. 2014. Data mining using R: learning with case studies (CRC) Press, Ed. Second. Minneapolis, Minnesota, USA. 87-165 pp. ISBN: 9781439810187.
Velázquez, L. N; Sasaki, Y.; Nakano, K.; Mejía, M. J y Romanchik, K. E. 2011. Detección de cenicilla en rosa usando procesamiento de imágenes por computadora. Revista Chapingo Serie Horticultura. 17(2):151-160. http://sourceforge.net/projects/opencvlibrary.
Wójtowicz, A.; Piekarczyk, J.; Czernecki, B. and Ratajkiewicz, H. 2021. A random forest model for the classification of wheat and rye leaf rust symptoms based on pure spectra at leaf scale. Journal of Photochemistry and Photobiology B. Biology. 223:1-11. https://doi.org/10.1016/j.jphotobiol.2021.112278.
Zhang, S.; Zhang, S.; Zhang, C.; Wang, X. and Shi, Y. 2019. Cucumber leaf disease identification with global pooling dilated convolutional neural network. 162:1-9. https://doi.org/10.1016/j.compag.2019.03.012.
Zhang, Y. and Wallace, B. 2015. A sensitivity analysis and practitioners’ guide to convolutional neural networks for sentence classification. 256-263 pp. http://arxiv.org/abs/1510.03820.
Zhao, Y. and Yang, L. 2023. Distance metric learning based on the class center and nearest neighbor relationship. Neural Networks. 164:631-644. https://doi.org/10.1016/j.neunet.2023.05.004.
Zapata, T. A.; Pérez, L. S. y Mora, F. J. 2014. Método basado en clasificadores k-NN parametrizados con algoritmos genéticos y la estimación de la reactancia para localización de fallas en sistemas de distribución. Revista Facultad de Ingeniería Universidad de Antioquia. 70:220-232. http://www.scielo.org.co/scielo.php?script=sci-arttext&pid=S012062302014000100021&lng=pt&tlng=es.
Descargas
Publicado
Cómo citar
Número
Sección
Licencia
Derechos de autor 2025 Revista Mexicana de Ciencias Agrícolas

Esta obra está bajo una licencia internacional Creative Commons Atribución-NoComercial 4.0.
Los autores(as) que publiquen en Revista Mexicana de Ciencias Agrícolas aceptan las siguientes condiciones:
De acuerdo con la legislación de derechos de autor, Revista Mexicana de Ciencias Agrícolas reconoce y respeta el derecho moral de los autores(as), así como la titularidad del derecho patrimonial, el cual será cedido a la revista para su difusión en acceso abierto.
Los autores(as) deben de pagar una cuota por recepción de artículos antes de pasar por dictamen editorial. En caso de que la colaboración sea aceptada, el autor debe de parar la traducción de su texto al inglés.
Todos los textos publicados por Revista Mexicana de Ciencias Agrícolas -sin excepción- se distribuyen amparados bajo la licencia Creative Commons 4.0 atribución-no comercial (CC BY-NC 4.0 internacional), que permite a terceros utilizar lo publicado siempre que mencionen la autoría del trabajo y a la primera publicación en esta revista.
Los autores/as pueden realizar otros acuerdos contractuales independientes y adicionales para la distribución no exclusiva de la versión del artículo publicado en Revista Mexicana de Ciencias Agrícolas (por ejemplo incluirlo en un repositorio institucional o darlo a conocer en otros medios en papel o electrónicos) siempre que indique clara y explícitamente que el trabajo se publicó por primera vez en Revista Mexicana de Ciencias Agrícolas.
Para todo lo anterior, los autores(as) deben remitir el formato de carta-cesión de la propiedad de los derechos de la primera publicación debidamente requisitado y firmado por los autores(as). Este formato debe ser remitido en archivo PDF al correo: revista_atm@yahoo.com.mx; revistaagricola@inifap.gob.mx.
Esta obra está bajo una licencia de Creative Commons Reconocimiento-No Comercial 4.0 Internacional.


